种质库在植物线粒体基因组替代速率异质性研究中取得重要进展
来源: 作者:刘芳,樊维姝 2020-06-22 浏览次数:
遗传多样性演化是进化基因组核心问题之一。被子植物线粒体基因组在大小、结构、基因及内含子数量等方面均具有多样性,这一特征使得线粒体基因组成为研究基因组复杂性的理想系统。通常认为植物线粒体基因组的进化速度较叶绿体基因组、核基因组低,且同一基因组内基因之间的进化速率较为一致。但前期通过对部分类群如匍匐筋骨草(Ajuga reptans)线粒体基因组的研究发现,基因间同义替代速率可相差2个数量级以上,其演化路径与成因尚不清楚。
中国科学院昆明植物研究所植物多样性与基因组学团队朱安丹研究组和李德铢研究组立足国家重大科技基础设施中国西南野生生物种质资源库,对唇形科(Lamiaceae)筋骨草亚科(Ajugoideae)全部四个主要分支中的9个属19个种的线粒体基因组进行了深入系统的分析,对线粒体基因组中出现的替代速率异质性的发生时间和相关机制进行了解析。研究发现,筋骨草亚科线粒体基因组中同义替代速率(dS)普遍高于非同义替代速率(dN),且同一基因组内不同基因出现加速的时间不同:有些基因的加速出现在筋骨草亚科的核心类群(core Ajugoideae)中(如rps12等),有些出现在筋骨草属及其姐妹属莸属等类群中(如atp6等),有些只出现在筋骨草属(Ajuga)中(如ccmC等),但几种模式中替代速率都在筋骨草属达到最大。进一步研究发现,替代速率变化最快的基因多数都位于基因簇上,而大部分这些基因簇在筋骨草属和其姐妹属中都是新形成的,新基因簇的形成要经历古老基因簇的断裂和新基因簇的融合,因此推测在这一过程中容易引入突变而导致基因突变速率提高。由于基因组的重组和基因转换会有一定的GC偏好性,因此通过对线粒体蛋白编码基因的第三位密码子中GC(GC3)含量的研究发现筋骨草属中几乎所有快速进化基因的GC3水平都有显著提高,且GC3含量的提高与dS显著相关。替代速率的提高还可能受到转录介导的修复或转录后修饰等因素的影响,具体可以表现在RNA编辑位点的丢失等方面,本研究通过结合转录组数据分析发现快速进化的基因丢失了所有祖先的RNA编辑位点,而中速和低速进化的基因只丢失了少量祖先的RNA编辑位点。该研究解析了植物线粒体基因组替代速率多样性,推测了线粒体基因组内同义替代速率异质性产生的机制,为植物线粒体基因组复杂性演化的深入研究提供了理论支持。
该研究成果以“Episodic and GC-biased bursts of intragenomic and interspecific synonymous divergence in Ajugoideae (Lamiaceae) mitogenomes”为题近日在线发表于植物科学领域主流杂志New Phytologist。中国科学院昆明植物研究所刘芳博士研究生和樊维姝博士为共同第一作者,李德铢研究员和朱安丹研究员为共同通讯作者。昆明植物所分子生物学实验中心杨俊波正高级工程师、中科院东亚植物多样性与生物地理学重点实验室向春雷博士、以及美国内布拉斯加大学林肯分校Jeffrey Mower博士也参与了该工作。该研究得到了中国科学院重大科技基础设施开放研究项目(No. 2017-LSF-GBOWS-02)、云南省基础研究计划(2019FD055)和种质库开放项目的支持。
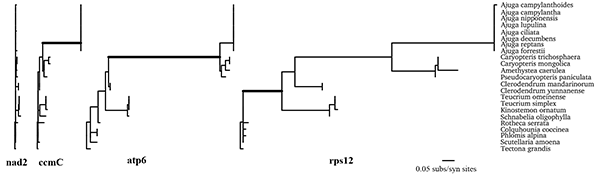
图1 不同线粒体编码基因发生替代速率加速的时间不同
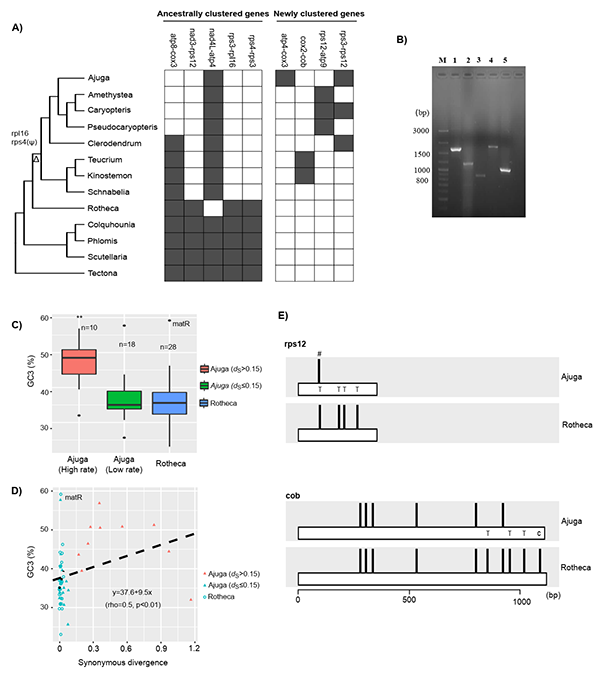
图2 基因替代速率与基因簇、GC3以及RNA编辑的关系